微生物无痕敲除技术介绍
发布时间:2019-07-26 15:00 | 点击次数:
转载自公众号:北京安必奇生物科技有限公司
基因敲除是一种常见的生物工程技术,可用于靶向基因组改造。有痕敲除的含义是每次敲除基因后都会在基因组中引入一个筛选标记。一方面,当进行连续敲除时,多种抗性标记在同一株菌体中堆叠,使得后面抗体标记的选择越来越困难。另一方面,抗体基因的插入可能会影响上下游基因的表达。因此,无痕(markerless/scarless)敲除越来越受到追捧,成为人们研究的重点。
无痕敲除技术
1 同源重组
同源重组技术包括Rec A和Red重组系统,Red系统具有明显的优势,包括同源臂较短,重组率较高等,被广泛用于大肠杆菌的基因组编辑。然后Red系统存在外源片段残留的情况,比如残留一个FRT重组酶作用位点。研究人员通过持续的研究,对该系统进行了持续的优化和改进,并开发出了无痕敲除技术,方便进行连续多个基因的敲除操作。下面我们将对这些优化策略进行解读。
1.1 环状质粒型同源重组载体
环状质粒载体是实现大肠杆菌基因敲除的经典策略,依靠的是RecA重组系统。RecA系统主要包括RecA、RecBCD、RuvA、RuvB、RuvC等。RecA具有与重组相关的活性,RecBCD可降解线性DNA片段,通过Chi序列识别靶位点,形成单链区域,然后再RecFOR、RecQ、RuvABC、SSB等蛋白共同作用下由RecA进行同源重组。
应用此类载体进行的基因敲除可分为2种类型,一种是通过同源单交换的插入型敲除,另一种是通过同源双交换的置换型敲除,后者可实现目标基因无痕敲除,但是第二步的效率较低。
1.2 线性双链DNA型同源重组载体
该载体依据Red重组系统。线性载体直接利用PCR的方法获得带有与靶基因同源的DNA片段和抗性基因,在Red重组系统的辅助下实现大肠杆菌的无痕敲除。相比于质粒型载体,该方法可以避免目标基因突变体(如基因点突变、基因缺失突变、基因插入突变)克隆和质粒载体的构建,简化了流程。但是该方法有一个缺点,即线性DNA容易被RecBCD酶降解。但这个问题可以被克服,Red重组系统设计时就已经包含了能抑制RecBCD对线性DNA降解的Gam蛋白。该载体同样适用于RecA重组系统。
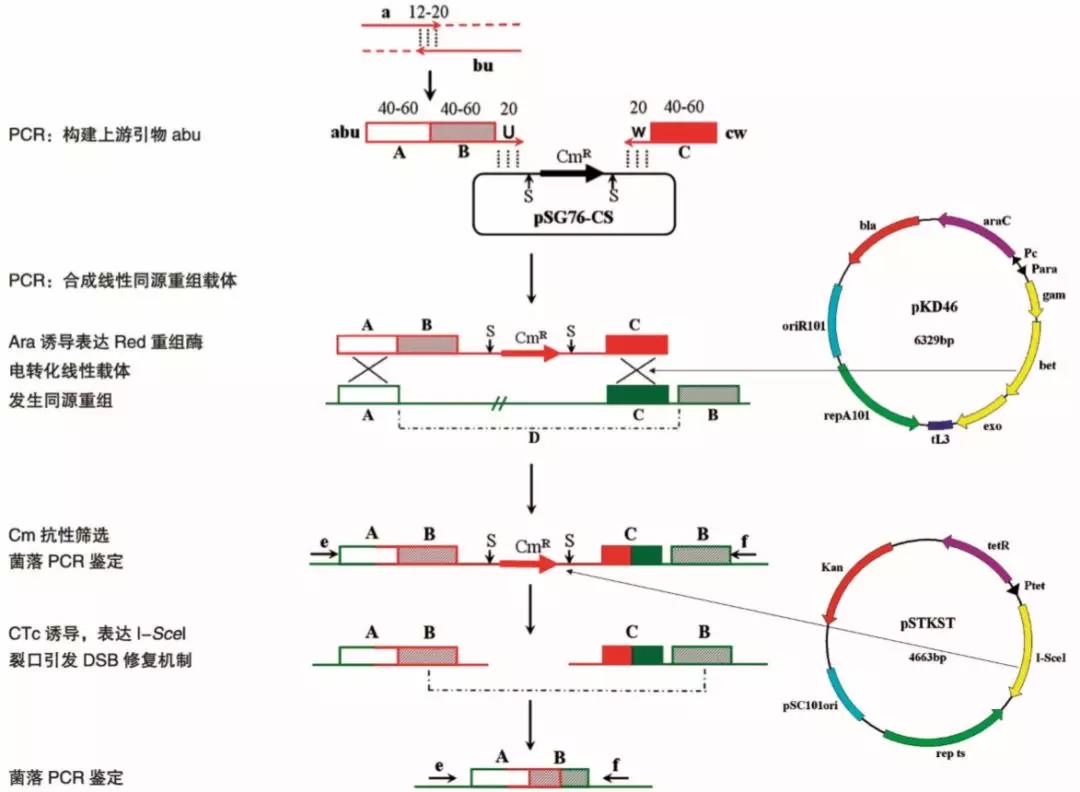
图1. 应用线性双链DNA型同源重组载体的无痕敲除技术原理图(Fehér等2008)
下图是葛高顺等人改进的大肠杆菌无痕敲除策略,第一步是采用两次PCR扩增出与目的基因两端同源并包含核酸内切酶I-SceI序列的氯霉素抗性片段。然后诱导质粒pKD46表达Red同源重组系统,并进行同源交换敲除目的基因,第二步进行无痕化处理。

图2. 质粒型无痕化基因敲除策略(葛高顺等2014)。
2 Cre/loxp系统
Cre重组酶于1982年在P1噬菌体中被发现,全长1029bp,能介导2个loxp位点间的基因删除或者倒立。loxp是一段回文序列,长约34bp,包含2个13bp的反向重复序列和1个8bp的不对称间隔区,是Cre酶的特异性识别位点。近年来,Cre/loxp位点特异性重组系统被用于无痕敲除。Herrmann等(2012)对该系统进行了改进,使得敲除率可达100%。作者先将2个loxp位点通过单交换的方法整合到敲除基因两侧,依靠Cre重组酶作用实现靶基因敲除。

图3. 改进后的Cre/loxp系统用于无痕敲除原理示意图(Herrmann等2012)。
3 CRISPR/Cas9系统
CRIPSR/Cas9技术是由RNA指导Cas9蛋白进行的定向基因组编辑技术,具有结构简单、安全性高、切点精准、价格低廉、可逆性、可同时编辑多个基因等优点,广泛应用于不同微生物中。这里主要讲一下CRIPSR无痕编辑。通过讲Red同源重组和CRISPR技术结合,可以实现CRIPSR无痕编辑,利用了Red重组酶的高效重组的优点和CRISPR系统识别并切割特异位点的优点。Reisch等(2015)发明了no-SCAR方法,能一步实现多个位点的无痕编辑(包括缺失、点突变、小片段插入等)。该方法需要构建2个质粒,包括受PTET启动子控制表达的cas9和tetR组成表达的质粒pCas9cr4和由受PTET启动子控制表达的sgRNA和受阿拉伯糖诱导启动子ParaB控制的λ-Red系统的质粒pKDsg-xxx。Ronda等(2016)基于MAGE(Multiplex Automated Genome Engineering)技术,创造了一种更为高效的CRMAGE(CRISPR/Cas9 and λ Red recombineering based MAGE technology),该方法对3个靶标的重组率高达96.5%-99.7%。

图4. no-SCAR系统用于大肠杆菌的基因组编辑(Reisch&Prather 2015)。

图5. CRMAGE系统的两个质粒结构(Ronda等 2016)。
参考文献:
1. Fehér T, Karcagi I, Győrfy Z, et al. Scarless engineering of the Escherichia coli genome[M]//Microbial Gene Essentiality: Protocols and Bioinformatics. Humana Press, 2008: 251-259.
2. 葛高顺, 张立超, 赵昕, 等. 大肠杆菌基因组基因无痕敲除的优化方法[J]. 中国生物工程杂志, 2014, 34(06): 68-74.
3. Herrmann S, Siegl T, Luzhetska M, et al. Site-specific recombination strategies for engineering actinomycete genomes[J]. Appl. Environ. Microbiol., 2012, 78(6): 1804-1812.
4. 谷燕燕, 耿伟涛, 宋存江. 链霉菌无痕敲除方法研究进展[J]. 生物工程学报, 2013, 29(8): 1100-1112.
5. 何庆,王茜,朱清华,方宏清.基因组工程在微生物细胞工厂构建中的应用进展[J].生物产业技术,2017(01):31-36.
6. Reisch C R, Prather K L J. The no-SCAR (Scarless Cas9 Assisted Recombineering) system for genome editing in Escherichia coli[J]. Scientific reports, 2015, 5: 15096.
7. Ronda C, Pedersen L E, Sommer M O A, et al. CRMAGE: CRISPR optimized mage recombineering[J]. Scientific reports, 2016, 6: 19452.






![T5]$60MG7WV8B]4)R5})5B9.png](https://img1.dxycdn.com/2017/0911/705/3232213446021413953-14.png!wh200)


